EL PARADIGMA DE SALMONELLA: Resistencias antimicrobianas
Escrito por:
Rafael J. Astorga Márquez1, Santiago Vega García2
1Departamento de Sanidad Animal. Facultad de Veterinaria de Córdoba (sa1asmar@uco.es)
2Departamento de Sanidad Animal. Facultad de Veterinaria. Univ. Cardenal Herrera-CEU Valencia
Medidas generales de control en salmonelosis
Debido a la amplia difusión de las salmonelosis y a la situación epidemiológica de estas infecciones/enfermedades, en la actualidad la erradicación total de las mismas no puede considerase como una estrategia de lucha. En cambio, si son factibles estrategias de control encaminadas a minimizar su prevalencia en las granjas animales y así reducir la presencia en la cadena alimentaria. Al considerarse enfermedades factoriales, deben implementarse medidas que controlen los factores de riesgo de tipo ambiental (protocolos L+DDD, gestión de residuos y cadáveres, control de calidad de agua y piensos, etcétera), así como los factores relacionados con el hospedador reforzando su resistencia (inmunoprofilaxis, metafilaxis y/o tratamientos específicos).
El uso de antimicrobianos (AMS) en la Unión Europea está limitado exclusivamente para el tratamiento de procesos con sintomatología clínica, quedando prohibido su utilización de forma preventiva y, en consecuencia, no deberán ser utilizados en el contexto de la estrategia de control de Salmonella, que en la mayoría de las ocasiones cursa como una infección subclínica. El uso indiscriminado de antimicrobianos puede potenciar la aparición de más resistencias en Salmonella puesto que es un patógeno frecuente en muchas explotaciones animales (p. ej. porcinas). Además, es posible que bajo la acción de determinados antimicrobianos se produzca lisis bacteriana masiva y liberación de endotoxina; en estos casos, y ante una endotoxemia inducida está indicado el uso de Antiinflamatorios No Esteroideos (AINEs).
En cualquier caso, es de carácter obligado realizar pruebas de sensibilidad in vitro en el laboratorio a partir de las cepas aisladas en los brotes clínicos (antibiogramas, e-test, Concentración Mínima Inhibitoria –CMI-). En estos casos los ensayos deben incluir los siguientes fármacos: ampicilina, cefalotina, ceftriaxona, cefotaxime, estreptomicina, gentamicina, ácido nalidíxico, enrofloxacina, ciprofloxacina, cloranfenicol, tetraciclina, sulfonamidas y trimetoprim-sulfametoxazol. La información de estas pruebas es muy válida para comprobar la lectura de la sensibilidad (S), así como los perfiles de resistencia (R-AMS).
En referencia al tratamiento sintomático, en los procesos salmonelósicos agudos es fundamental controlar el fallo cardiorrespiratorio y hemodinámico a base de cardiotónicos y analépticos cardiorrespiratorios (p. ej. efedrina, etilefrina, teofilina), así como con sustancias vasopresoras (p. ej. dopamina, noradrenalina) En los procesos gastroentéricos graves, se producen desviaciones bioquímicas consistentes en desequilibrio ácido-base y deshidratación que sitúan a los animales al borde del shock. En estos casos debemos plantear diversas estrategias de actuación: (i) fluidoterapia inicialmente con soluciones isotónicas y posteriormente glucosadas (ii) supresión láctea, ya que altera los fenómenos de absorción, (iii) aporte nutritivo vía parenteral (glúcidos, vitaminas, aminoácidos); (iv) uso de probióticos (p. ej. Lactobacillus spp.) para la reposición de la microbiota intestinal.
Antimicrobianos y resistencia
La Organización Mundial de la Salud (OMS) ha alertado desde hace años del uso inapropiado de los antimicrobianos en las granjas animales, así como del importante papel epidemiológico de emergencia y diseminación de cepas resistentes con alto impacto en la salud pública e industria alimentaria (Figuras 1-2).
Los últimos documentos publicados por la Agencia Europea de Seguridad Alimentaria y el Centro Europeo para el Control de Enfermedades (EFSA y ECDC, 2017) sobre «Resistencia antimicrobiana en bacterias zoonóticas», señalan la amplia difusión de cepas multirresistentes (MDR, del inglés Multiple Drug Resistance) de Salmonella spp. detectadas con altos niveles de resistencia frente a enrofloxacina, ceftiofur y trimetoprim-sulfametoxazol.
Figuras 1-2. Divulgación de la OMS sobre resistencia antimicrobiana.


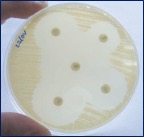
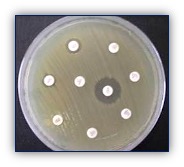
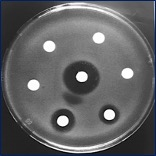
Por todo ello, se ha hecho imprescindible establecer medidas de vigilancia epidemiológica para detectar el desarrollo de resistencias antibacterianas frente a determinados patógenos, entre ellos Salmonella spp., sobre todo en porcinocultura y avicultura. En este sentido, Astorga et al. (2007) detectaron en el sur de España y en cerdos de cría intensiva (121 granjas analizadas) prevalencias del 33%, siendo Rissen, Derby y Typhimurium los serotipos más frecuentes, y DT104 el fagotipo más prevalente en cepas ST. Los niveles de multirresistencia fueron del 64%, con elevados porcentajes de resistencia frente a ampicilina, estreptomicina, sulfonamidas y tetraciclinas (patrón R-ASSuT) (Figura 3-4). Además, Gómez-Laguna et al. (2011) detectaron en cerdo Ibérico una prevalencia de infección por Salmonella spp. en las granjas muestreadas (67 granjas/804 animales), idéntica a la observada previamente en sistemas intensivos en la misma zona geográfica; además, se detectaron serotipos poco comunes de origen silvestre (ej. Mikawasima, Hessarek) y una mayor diversidad de fagotipos. En este caso, el 58% de las cepas mostró resistencia frente a uno o más antimicrobianos siendo los niveles de cepas MDR inferiores a los encontrados en cerdo de capa blanca (36% versus 64%).
Figuras 3-4. Ensayos de sensibilidad in vitro mediante difusión de discos antimicrobianos en agar Müeller-Hinton. Se observa plena sensibilidad (izquierda) y multirresistencia (derecha).
 .
. 
La búsqueda de nuevos métodos de control frente a este microorganismo principal indicador de zoonosis es un objetivo prioritario de numerosas investigaciones. En este sentido, nuestro grupo ha investigado los efectos sinérgicos in vitro de ciertos antimicrobianos (enrofloxacina, ceftiofur y Sxt) junto a determinados aceites esenciales (EOs) (canela, clavo, orégano, tomillo) sobre cepas MDR de Salmonella entérica (Solarte et al., 2017, 2018), obteniendo resultados muy satisfactorios que auguran nuevas estrategias de control.
En la especie humana, algunos cuadros clínicos asociados a cepas de Salmonella Enteritidis (SE), S. Typhimurium (ST) o Variante monofásica de S. Typhimurium (cepas mST), son severos y requieren hospitalización (EFSA, 2010). El control de estas infecciones requiere terapia antimicrobiana a base de fluoroquinolonas o ceftriaxona, administrada esta última a niños para evitar el daño de cartílago frecuentemente asociado al uso de quinolonas (Foley y Lynne, 2008).
Los patrones de resistencia de estos serotipos, incluida la variante monofásica mST, pueden variar desde un 100% de sensibilidad a multirresistencia. A pesar de que los niveles de resistencia de S. Typhimurium han ido descendiendo en varios países de Europa, la incidencia de cepas mST (S. 4,[5],12:i:-) resistentes parece ir «in crescendo» (Switt et al., 2009). Por todo ello, existe una preocupación a nivel mundial por el aumento de la frecuencia de presentación de cepas mST de origen animal con carácter MDR, y que pueden potencialmente ingresar en la cadena alimentaria y afectar al ser humano.
Un hecho particularmente relevante en las cepas monofásicas es que la mayoría de los aislamientos virulentos exhiben resistencia mediada por plásmidos a una amplia gama de antimicrobianos. De forma similar a su linaje ancestral <Salmonella Typhimurium DT104>, las cepas mST expresan frecuentemente una múltiple resistencia frente a ampicilina, estreptomicina, sulfonamida y tetraciclina. Este perfil de resistencia (R-ASSuT) está codificado cromosómicamente (Hopkins et al., 2010).
La resistencia antimicrobiana de Salmonella puede deberse a varios determinantes localizados en el cromosoma bacteriano o en plásmidos (Foley y Lynne, 2008; Switt et al., 2009). Estos «determinantes genéticos» pueden ser responsables de la expresión de mecanismos de resistencia intrínsecos relacionados con la producción de beta-lactamasas, modificación de la composición antimicrobiana por enzimas bacterinas, variaciones de la permeabilidad bacteriana, presencia de bombas de flujo (efflux pumps), o modificaciones de receptores diana (Foley y Lynne, 2008).
La resistencia antimicrobiana puede producirse también a través de la expresión de mecanismos de resistencia adquirida, que emergen por medio de mutaciones puntuales en genes cromosomiales, o a través de la adquisición de elementos móviles tales como plásmidos, transposones o islas genómicas (Switt et al., 2009). La transferencia de resistencia se puede producir directamente de la misma o de diferentes especies o géneros de bacterias, o indirectamente a través del medio ambiente (Foley y Lynne, 2008; EFSA, 2010). La microbiota intestinal de los seres humanos y animales está expuesta a menudo a compuestos antimicrobianos de diferentes clases, concentraciones y frecuencias de exposición, utilizados para la terapia, la profilaxis o metafilaxis. Esta exposición puede derivar de los piensos o del entorno (Martins da Costa et al., 2007).
La aparición, selección y diseminación de bacterias resistentes a los antimicrobianos se siguen atribuyendo principalmente a la presión selectiva del mal uso y abuso de antibióticos (Sayah et al., 2005), de este modo las bacterias intestinales pueden volverse resistentes a algunos compuestos antimicrobianos y por lo tanto transmitir esta resistencia a Salmonella, que ocupa el mismo nicho ecológico.
En 1997 se identificaron por vez primera en España cepas de S. 4,[5],12:i:- con carácter MDR (Guerra et al., 2001) (Tabla 1). Como ya citamos anteriormente, el patrón de resistencia más frecuente en estas cepas es R-ASSuT, detectado en el 30% de las infecciones de origen humano y en granjas animales en los primeros años de aislamientos (EFSA, 2010; Lucarelli et al., 2010). Este patrón emergió en Italia en el año 2000 para extenderse luego por Dinamarca, Reino Unido, Estados Unidos (EE. UU.), España, Francia y República Checa (Lucarelli et al., 2010). Los genes responsables de este fenotipo MDR se encuentran presentes en una isla genómica que usualmente incluye a blaTEM, strA-strB, sul2 y tet(B) (Hauser et al., 2010; Lucarelli et al., 2010).
Tabla 1. Cronología de aislamientos de cepas mST (S. 4,5,12; i; -)
(Adaptado de EFSA 2010).

Andrés-Barranco et al. (2016) describieron la circulación de cepas monofásicas aisladas a partir de animales y ambiente en granjas porcinas, aves silvestres y roedores en el norte de España, con un marcado carácter multirresistente. En este trabajo, el 85,8% de las cepas fueron resistentes a uno o más antimicrobianos, con un 94,1% de cepas MDR. Desde el punto de vista fenotípico, el fagotipo más prevalente en estas cepas mST fue U311 (40,7%) y DT195 (22%). Este perfil fue más similiar a cepas europeas que al denominado «Spanish clon» y mostró un patrón de resistencia diferente al clásico R-ASSuT.
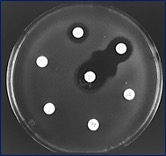
Los fenotipos MDR incluyen cepas S. 4,[5],12:i:- que albergan integrones de clase 1 o plásmidos de resistencia, resistentes a ampicilina, cloranfenicol, gentamicina, estreptomicina, sulfametoxazol, tetraciclina y trimetoprim. Estos rasgos de resistencia son debidos principalmente a la expresión de bla-TEM-1, que codifica para un amplio espectro de beta-lactamasas responsables de la resistencia a penicilinas y amino-penicilinas. Otros genes de resistencia detectados son: (i) blaCTX-M-1, para beta-lactamasas de espectro extendido (BLEE) (Figuras 5-6); (ii) cmlA1, para una bomba de flujo responsable de la resistencia frente al cloranfenicol; (iii) aac(3)-IV y aacA2, para enzimas que modifican los sitios activos de gentamicina y estreptomicina alterando la acción de estos fármacos; (iv) aadA1, sul1 y sul2, para enzimas responsables de la resistencia a sulfonamidas; (v) sul3 y tet(A), para un mecanismo de bomba de flujo responsable de la resistencia a tetraciclina; (vi) dfrA12, para una enzima responsable de la resistencia a trimetoprim (Guerra et al., 2001; Foley y Lynne, 2008; Switt et al., 2009).
Figuras 5-6. Principales betalactamasas: (izquierda) AMPc; (derecha) CTX-M-1 para BLEE.
Recientemente, el Ministerio de Sanidad, Consumo y Bienestar Social junto a la Agencia Española de Medicamentos y Productos Sanitarios (AEMPS) han publicado el nuevo Plan Nacional de Resistencia a los Antibióticos (PRAN, 2019-2021). En este nuevo documento se revisan los siguientes aspectos: (i) vigilancia del consumo de antibióticos y la resistencia; (ii) control de la resistencia a los antibióticos; (iii) prevención de la necesidad del uso de antibióticos; (iv) estrategia común sobre la investigación; (v) formación; (vi) comunicación y sensibilización de la población; (vii) medio ambiente. En este nuevo plan se hace especial énfasis en la participación y colaboración de todos los profesionales sanitarios para frenar las resistencias a los antibióticos (Figura 7).
Figura 7. Infografía del Plan Nacional de Resistencia a los Antibióticos. ¿Qué espera el PRAN de ti? (Fuente: PRAN, 2019-2021)

Por su parte, y muy recientemente, la European Medicines Agency (EMA, Science Medicine Health), ha editado el documento «categorización de antimicrobianos para uso prudente y responsable en animales». En esta infografía (Figura 8), se clasifican los grupos de antimicrobianos según cuatro categorías o niveles: (A) prohibidos; (B) restricciones (C) precaución; (D) prudencia. Asimismo, se reflejan ejemplos de sustancias autorizadas para uso médico y veterinario en la Unión Europea.
Figura 8. Categorización de antimicrobianos para uso prudente y responsable en veterinaria
(Fuente: EMA, 2020).

Referencias bibliográficas
- Andrés-Barranco, S., Vico, J.P., Marín, C.M., Herrera-León, S. Mainar-Jaime, R.C. (2016). Characterization of Salmonella enterica Serovar Typhimurium Isolates from Pigs and Pig Environment-Related Sources and Evidence of New Circulating Monophasic Strains in Spain. Journal of Food Protection. 79 (3): 407-12.
- Astorga Márquez, Rafael J., Aurora Echeita Salaberria, Alfonso Maldonado García, Silvia Valdezate Jiménez, Alfonso Carbonero Martínez, Ana Aladueña García, Antonio Arenas Casas. (2007). Surveillance and antimicrobial resistance of Salmonella strains isolated from slaughtered pigs in Spain. Journal of Food Protection. 70(6): 1502-1506.
- EFSA Panel on Biological Hazards (BIOHAZ). (2010). Scientific Opinion on monitoring and assessment of the public health risk of “Salmonella Typhimurium-like” strains. EFSA Journal. 8, 10, (Oct 2010), pp. 1-48.
- Foley, S.L., and Lynne, A.M. (2008). Food animal-associated Salmonella challenges: Pathogenicity and antimicrobial resistance. Journal of Animal Science, 86, 14, (Apr2008), pp. E173–E187, ISSN 0021-8812.
- Gómez-Laguna, Jaime., Manuela Hernández, Eva Creus, Aurora Echeita, Julio Otal, Silvia Herrera-León, Rafael J. Astorga. (2011). Prevalence and antimicrobial susceptibility of Salmonella infections in free-range pigs. Veterinary Journal. 190: 176-178.
- Guerra, B., Soto, S. A., Argüelles, J. M. Mendoza, C. M. (2001). Multidrug resistance is mediated by large plasmids carrying a class 1 integron in the emergent Salmonella enterica serotype (4,5,12:i-). Antimicrobial Agents and Chemotherapy. 45: 1305-1308.
- Hauser E., et al. (2010). Pork contaminated with Salmonella enterica serovar 4,[5],12:i:-, an emerging health risk for humans. Appl Environ Microbiol. 14: 4601-4610.
- Hopkins, K.L., Kirchner, M., Guerra, B., Granier, S.A., Lucarelli, C., Porrero, M.C., Jakubczak, A., Threlfall, E.J., Mevius, D.J. (2010). Multiresistant Salmonella enterica serovar 4, [5],12:i:- in Europe: a new pandemic strain?. Eurosurveillance, 15, 22, (Jun 2010), pp. 1-9.
- Lucarelli, C., Dionisi, A.M., Torpdahl, M., Villa, L., Graziani, C., Hopkins, K., Threlfall, J., Caprioli, A., Luzzi, I. (2010). Evidence for a second genomic island conferring multidrug resistance in a clonal group of strains of Salmonella enterica serovar Typhimurium and its monophasic variant circulating in Italy, Denmark, and the United Kingdom. Journal of Clinical Microbiology, 48, 6, (Jun 2010), pp. 2103-2109, ISSN: 0095-1137.
- Martins da Costa, P., Oliveira, M., Bica, A., Vaz-Pires, P., Bernardo, F. (2007). Antimicrobial resistance in Enterococcus and Escherichia coli isolated from poultry feed and feed ingredients. Veterinary Microbiology, 120, 1-2, (Feb 2007), pp. 122–131.
- Sayah, R.S., Kaneene, J.B., Johnson, Y., Miller, R. (2005). Patterns of antimicrobial resistance observed in Escherichia coli isolates obtained from domestic- and wild animal fecal samples, human septage, and surface water. Applied and Environmental Microbiology, 71, 3, (Mar 2005), pp. 1394–1404, ISSN 0099-2240.
- Solarte, Ana Lucía., Rafael Jesús Astorga, Fabiana Aguiar, Ángela Galán-Relaño, Alfonso Maldonado, and Belén Huerta. (2017). Combination of Antimicrobials and Essential Oils as an Alternative for the Control of Salmonella enterica Multiresistant Strains Related to Foodborne Disease. Foodborne Pathogens and Disease. 14 (10): 558-563.
- Solarte, Ana Lucía., Rafael Jesús Astorga, Fabiana Carolina de Aguiar, Cristina De Frutos, Belén Barrero-Domínguez, and Belén Huerta. (2018). Susceptibility Distribution to Essential Oils of Salmonella enterica Strains Involved in Animal and Public Health and Comparison of the Typhimurium and Enteritidis Serotypes. Journal of Medicinal Food. 21 (9): 946-950.
- Switt, A.I.M., Soyer, Y., Warnick, L.D., Wiedmann, M. (2009). Emergence, distribution and molecular and phenotypic characteristics of Salmonella enterica serotype 4,5,12:i:-. Foodborne Pathogens and Disease, (May 2009), pp. 407–415, ISSN 1535-3141.
- The European Union summary report on antimicrobial resistance in zoonotic and indicator bacteria from humans, animals and food in 2017. EFSA Journal 2019; 17 (2): 5598.
https://amazingbooks.es/el-paradigma-salmonella